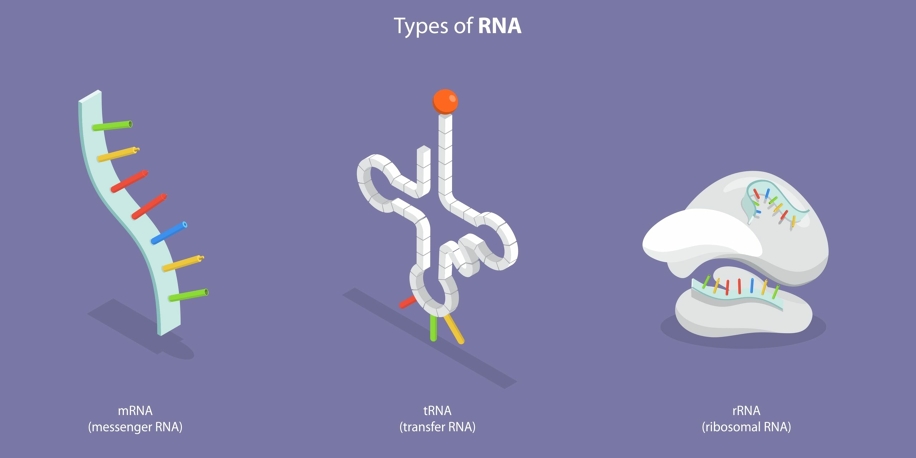
L’RNA, o acido ribonucleico, è cruciale per il funzionamento delle nostre cellule. La sua funzione più conosciuta è quella di RNA messaggero (mRNA): prende le informazioni contenute nel DNA, che si trova nel nucleo, e le porta nel citoplasma, dove vengono usate per costruire le proteine grazie ai ribosomi. Ma l’RNA non fa solo questo. Esistono anche altri tipi di RNA, come l’RNA ribosomiale (rRNA), che fa parte della struttura dei ribosomi, e l’RNA di trasporto (tRNA), che aiuta ad assemblare gli amminoacidi nell’ordine giusto per formare una proteina.
Oggi sappiamo che l’RNA ha molte altre funzioni, e probabilmente ne scopriremo di nuove in futuro. Questo ci aiuta a capire anche perché gran parte del nostro DNA non serve direttamente a creare proteine, ma ha comunque un ruolo importante per far funzionare correttamente la cellula e garantire la nostra sopravvivenza.
La struttura tridimensionale dell’RNA
Siamo abituati a pensare all’RNA come una semplice sequenza lineare di nucleotidi, che fornisce informazione come una parola in un testo. In realtà l’RNA può anche ripiegarsi su sé stesso formando complesse strutture tridimensionali, e proprio queste strutture sono fondamentali per molte delle sue funzioni.
L’esempio più lampante sono l’rRNA e il tRNA, che riescono a svolgere il loro ruolo grazie proprio alla loro forma. Ma non sono gli unici: esistono molti altri tipi di RNA con strutture 3D complesse essenziali per il corretto funzionamento della cellula.
Queste strutture sono state osservate anche nei virus, che spesso hanno un genoma costituito da RNA. È il caso di virus come SARS-CoV-2, quello dell’influenza e molti altri. Dato che i virus devono condensare tante informazioni in poco spazio, sfruttano al massimo l’RNA: non solo per il suo contenuto genetico, ma anche per le forme tridimensionali che può assumere.
Alcuni virus possiedono, all’inizio del loro RNA, delle strutture particolari chiamate IRES (siti interni di riconoscimento del ribosoma). Si tratta di complesse strutture tridimensionali capaci di legarsi direttamente al ribosoma e ad altre proteine coinvolte nella sintesi delle proteine, anche quando manca il classico ‘cappuccio’ (CAP) che normalmente segnala l’inizio della traduzione. Anche alcuni mRNA umani contengono delle IRES per poter produrre proteine in condizioni di stress.
Altri virus utilizzano strutture 3D per impacchettare correttamente il loro RNA alla fine del ciclo di replicazione, in modo da prepararlo per l’infezione di nuove cellule. Ci sono anche virus che sfruttano queste strutture per far ‘slittare’ il ribosoma lungo l’RNA in modo controllato: questo permette di leggere lo stesso tratto di RNA in modi diversi, producendo così quantità diverse di proteine da una singola sequenza. Questo meccanismo si chiama spostamento programmato del ribosoma e si osserva, ad esempio, nei coronavirus e nel virus dell’HIV (virus dell’immunodeficienza acquisita).
L’RNA come strumento di editing genetico
L’esempio più famoso dell’uso dell’RNA come strumento per modificare il DNA è legato alla scoperta del sistema CRISPR-Cas9, che ha valso il premio Nobel per la chimica a Jennifer Doudna ed Emmanuelle Charpentier nel 2020. Questo meccanismo è stato scoperto nei batteri, dove funziona come un sistema di difesa contro i virus.
L’enzima Cas9 è in grado di tagliare il DNA dei virus in punti specifici, guidato da una molecola di RNA chiamata ‘RNA guida‘. Questo RNA deriva da particolari sequenze ripetute nel DNA batterico chiamate CRISPR, che funzionano come una sorta di archivio dei virus incontrati in passato.
I ricercatori hanno adattato questo sistema naturale per usarlo come uno strumento di editing genetico: oggi può essere programmato per tagliare il DNA in punti precisi, con l’obiettivo di correggere o disattivare geni difettosi. Viene utilizzato sia nella ricerca scientifica, che in ambito medico, ad esempio per curare alcune malattie genetiche come l’anemia falciforme o altre patologie, per la quale sono attualmente in corso studi clinici.
Altri esempi di RNA che regolano l’espressione dei geni sono i microRNA (miRNA) e gli small interfering RNA (siRNA). Entrambi agiscono sul mRNA, bloccandone la funzione o promuovendone la degradazione, anche se lo fanno con meccanismi leggermente diversi.
In entrambi i casi, questi piccoli RNA riescono a riconoscere l’mRNA bersaglio grazie alla complementarità della sequenza: i siRNA si legano in modo quasi perfetto, mentre i miRNA possono legarsi anche in modo parziale.
I miRNA sono prodotti naturalmente dalle nostre cellule e svolgono un ruolo fondamentale nello sviluppo, nella regolazione genica e nella risposta a stress o segnali ambientali. I siRNA, invece, sono stati scoperti inizialmente in piante e nel moscerino della frutta e oggi vengono usati come strumenti nella ricerca scientifica e nello sviluppo di nuove terapie.
Partendo da questi meccanismi di interferenza, oggi si stanno sviluppando diverse terapie innovative che agiscono proprio sull’RNA. Una di queste strategie prevede l’uso di sequenze chiamate ‘antisenso‘, che sono piccoli pezzi di RNA (o DNA) progettati per legarsi all’RNA messaggero e bloccarne la traduzione in proteina, oppure per favorirne la degradazione.
Questi antisenso vengono spesso modificati chimicamente, così da renderli più stabili e più efficaci, evitando che vengano rapidamente degradati all’interno della cellula. Le terapie basate su questa tecnologia si chiamano oligonucleotidi antisenso (ASO), e sono in fase di studio per il trattamento di diverse malattie.
Potremmo citare molti altri esempi che mostrano quanto l’RNA e le sue modificazioni siano cruciali per il corretto funzionamento delle cellule. Ma già questi casi, come CRISPR/Cas9 e gli ASO, ci fanno capire una cosa importante: per sviluppare nuove terapie è essenziale conoscere a fondo la biologia dell’RNA.
Attività da proporre in classe:
Realizzare un video stile reel su uno dei tipi di RNA e le sue funzioni:
Scegliere uno degli esempi di RNA citati e realizzare a gruppi un breve video in stile divulgativo sulla funzione dell’RNA
Trova l’RNA messaggero tra quelli proposti:
L’RNA messaggero contiene sempre una sequenza iniziale chiamato 5’ untranslated region seguita dal vero inizio della traduzione che corrisponde alla tripletta AUG che corrisponde all’amminoacido metionina. La sequenza codificante è poi di variabile lunghezza e al suo termine c’è un codone di stop (UAA, UAG o UGA) seguito da una sequenza non codificante, chiamata 3’ untranslated region e da una serie di adenine che garantiscono la stabilità dell’RNA.
(Le sequenze sono semplificate e non corrispondono a sequenze reali)
a) AAA CGA AUU AUG GCU ACU GGA AUG AAA CGA UAA CCA AAG UUA AAA AAA AAA
b) AAA CGA AUU AUU GCU ACU GGA AGG AAA CGA UAA CCA AAG UUA AAA AAA AAA
c) AAA CGA AUU AUG UAG ACU GGA AUG AAA CGA UAA CCA AAG UUA AAA AAA AAA
Soluzione:
La soluzione corretta è la A perché nella sequenza B non c’è un codone di inizio AUG, mentre nella sequenza C il codone di inizio è seguito da un codone di stop che impedisce di sintetizzare una proteina.
N.B. Sulla base di regole semplici come queste possiamo predire all’interno di genomi umani e di altre specie la presenza di geni, o le strutture secondarie dell’RNA o anche la presenza di sequenze che codificano per micro RNA.
